Kit de PCR Blood Direct
Recursos
■ Simples e rápido: a amplificação por PCR pode ser realizada diretamente usando sangue como molde, sem a necessidade de etapas tediosas de preparação de amostra e extração de DNA.
■ Alta pureza: pular as etapas de pré-tratamento da amostra e extração de DNA pode ajudar a evitar a contaminação cruzada das amostras.
■ Alta produtividade: A identificação de PCR para amostras em grande escala pode ser realizada combinando o kit com placas de PCR de 96/384 poços.
■ Universalidade forte: este kit pode amplificar com eficiência fragmentos de alto GC ou fragmentos com estrutura secundária complexa e o comprimento de amplificação pode ser de até 5 kb.
■ Forte resistência ao estresse: este kit pode ser aplicado a várias espécies e amostras de sangue preservadas de diferentes maneiras.
Formulários
Os produtos de PCR deste kit contêm “A” na extremidade 3′, que pode ser usado diretamente para a clonagem do vetor TA. Este kit pode ser usado para a amplificação de fragmentos de DNA genômico, análise genética de alto rendimento e análise de genotipagem (como detecção de gene).
Todos os produtos podem ser personalizados para ODM / OEM. Para detalhes,clique em Serviço personalizado (ODM / OEM)
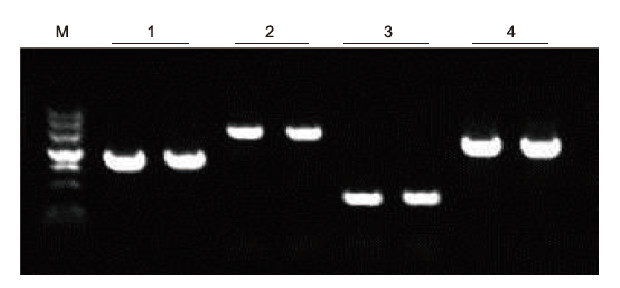 |
Usando anticoagulação EDTA humana como molde, 4 genes com diferentes conteúdos de GC foram amplificados pelo kit Blood Direct PCR. O sistema de reação de PCR foi de 20 μl, e 1 μl de sangue foi usado como molde. M: Marcador II de TIANGEN; 1: tamanho do fragmento 1090 bp, conteúdo de GC 68,1%; 2: tamanho do fragmento 1915 bp, conteúdo de GC 70,4%; 3: tamanho do fragmento 448 bp, conteúdo de GC 74,8%; 4: Tamanho do fragmento 1527 bp, conteúdo de GC 61,5%. Resultados experimentais: o kit Blood Direct PCR pode amplificar efetivamente fragmentos de DNA com conteúdo de GC na faixa de 61,5% -74,8%, sugerindo que ele é capaz de amplificar fragmentos de alto GC. |
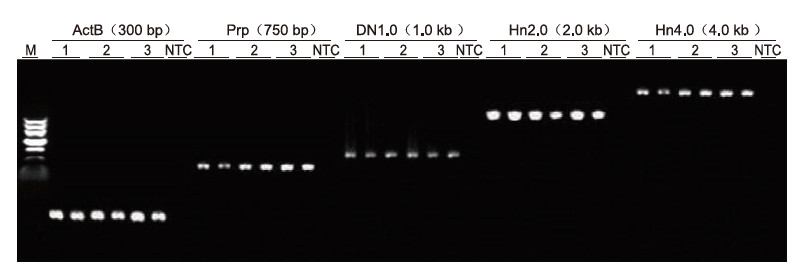 |
Usando anticoagulação EDTA humana como molde, 5 genes com diferentes comprimentos (ActB, Prp, DN1.0, Hn2.0 e Hn4.0) foram amplificados pelo kit Blood Direct PCR. O sistema de reação de PCR foi de 20 μl, e 1 μl de sangue foi usado como molde. M: Marcador II de TIANGEN; 1-3: 3 amostras de sangue diferentes; NTC: controle sem primers. Resultados experimentais: o kit Blood Direct PCR pode amplificar fragmentos com comprimento de até 4 kb, sugerindo que é capaz de amplificar fragmentos longos. |
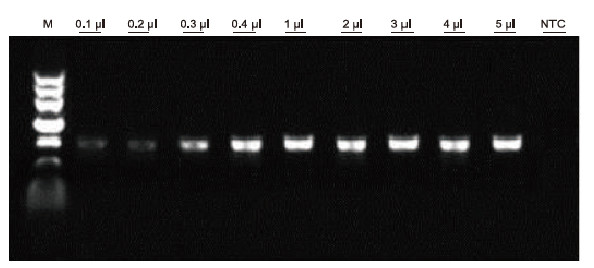 |
Usando anticoagulação EDTA humana como molde, o kit Blood Direct PCR foi usado para detecção por PCR de diferentes amostras de sangue. O sistema de reação de PCR foi de 20 μl, e 1 μl de sangue foi usado como molde. M: Marcador II de TIANGEN; 1-9: a quantidade de carga de sangue é 0,1 μl, 0,2 μl, 0,3 μl, 0,4 μl, 1 μl, 2 μl, 3 μl, 4 μl e 5 μl, respectivamente; NTC: controle sem modelo Resultados experimentais: O kit Blood Direct PCR tem forte resistência ao sangue e pode amplificar amostras de sangue com uma faixa de carga de 0,1-5 μl. |
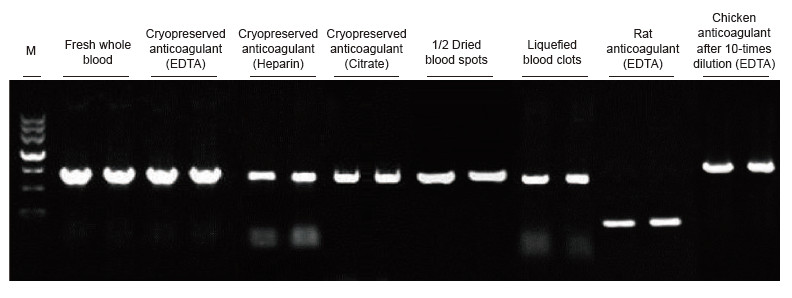 |
Amostras de sangue de humanos, ratos, galinhas e outras espécies com diferentes tratamentos foram usadas como modelos. O kit Blood Direct PCR foi usado para amplificar PRNP (humano, 750 pb), Actina (rato, 200 pb) e β-Actina (Galinha, 1,0 kb). O sistema de reação de PCR foi de 20 μl, e 1 μl de sangue foi usado como molde. M: Marcador II de TIANGEN. Resultados experimentais: O kit Blood Direct PCR pode ser aplicado em uma ampla gama de amostras, e a detecção direta por PCR pode ser realizada em amostras de sangue de várias espécies com diferentes tratamentos. |
Modelo A-1
■ O modelo contém impurezas de proteínas ou inibidores Taq, etc. —— Purifique o modelo de DNA, remova as impurezas de proteínas ou extraia o DNA do modelo com kits de purificação.
■ A desnaturação do modelo não está completa —— Aumente apropriadamente a temperatura de desnaturação e prolongue o tempo de desnaturação.
■ Degradação do modelo ——Reprepare o modelo.
A-2 Primer
■ Baixa qualidade dos primers —— Sintetize novamente o primer.
■ Degradação do primer —— Aliquote os primers de alta concentração em um pequeno volume para preservação. Evite congelamento e descongelamento múltiplo ou criopreservação de 4 ° C por longo prazo.
■ Projeto inadequado de primers (por exemplo, o comprimento do primer não é suficiente, dímero formado entre os primers, etc.) -Redesign primers (evita a formação de primer dímero e estrutura secundária)
A-3 Mg2+concentração
■ Mg2+ a concentração está muito baixa —— Aumente o Mg de maneira adequada2+ concentração: Otimize o Mg2+ concentração por uma série de reações de 1 mM a 3 mM com um intervalo de 0,5 mM para determinar o Mg ideal2+ concentração para cada molde e primer.
Temperatura de recozimento A-4
■ A alta temperatura de recozimento afeta a ligação do primer e do template. ——Reduza a temperatura de recozimento e otimize a condição com um gradiente de 2 ° C.
Tempo de extensão A-5
■ Tempo de extensão curto —— Aumente o tempo de extensão.
Fenômenos: Amostras negativas também mostram as bandas da sequência alvo.
Contaminação A-1 de PCR
■ Contaminação cruzada da sequência-alvo ou produtos de amplificação ——Cuidado para não pipetar a amostra que contém a sequência-alvo na amostra negativa ou derramar para fora do tubo de centrífuga. Os reagentes ou equipamentos devem ser autoclavados para eliminar os ácidos nucléicos existentes, e a existência de contaminação deve ser determinada por meio de experimentos de controle negativo.
■ Contaminação do reagente —— Aliquote os reagentes e armazene-os em temperatura baixa.
A-2 Primer
■ Mg2+ a concentração está muito baixa —— Aumente o Mg de maneira adequada2+ concentração: Otimize o Mg2+ concentração por uma série de reações de 1 mM a 3 mM com um intervalo de 0,5 mM para determinar o Mg ideal2+ concentração para cada molde e primer.
■ Projeto de primer impróprio e a sequência alvo tem homologia com a sequência não alvo. —— Refaça os primers.
Fenômenos: As bandas de amplificação de PCR são inconsistentes com o tamanho esperado, seja grande ou pequeno, ou às vezes ocorrem bandas de amplificação específicas e bandas de amplificação não específicas.
A-1 Primer
■ Baixa especificidade do primer
—— Primer de redesenho.
■ A concentração do primer está muito alta —— Aumente adequadamente a temperatura de desnaturação e prolongue o tempo de desnaturação.
A-2 Mg2+ concentração
■ O Mg2+ a concentração é muito alta —— Reduza adequadamente a concentração de Mg2 +: Otimize o Mg2+ concentração por uma série de reações de 1 mM a 3 mM com um intervalo de 0,5 mM para determinar o Mg ideal2+ concentração para cada molde e primer.
Polimerase A-3 Termostável
■ Quantidade excessiva de enzima - reduza a quantidade de enzima de forma adequada em intervalos de 0,5 U.
Temperatura de recozimento A-4
■ A temperatura de recozimento está muito baixa —— Aumente apropriadamente a temperatura de recozimento ou adote o método de recozimento de dois estágios
Ciclos de PCR A-5
■ Muitos ciclos de PCR —— Reduza o número de ciclos de PCR.
A-1 Primer—— Especificidade pobre —— Desenhe novamente o primer, mude a posição e o comprimento do primer para aumentar sua especificidade; ou realizar PCR aninhado.
DNA de modelo A-2
——O modelo não é puro —— Purifique o modelo ou extraia o DNA com kits de purificação.
A-3 Mg2+ concentração
——Mg2+ a concentração é muito alta —— Reduza o Mg de maneira adequada2+ concentração: Otimize o Mg2+ concentração por uma série de reações de 1 mM a 3 mM com um intervalo de 0,5 mM para determinar o Mg ideal2+ concentração para cada molde e primer.
A-4 dNTP
—— A concentração de dNTPs está muito alta —— Reduza a concentração de dNTP de forma adequada
Temperatura de recozimento A-5
—— Temperatura de recozimento muito baixa —— Aumente apropriadamente a temperatura de recozimento
Ciclos A-6
—— Muitos ciclos —— Otimize o número do ciclo

O primeiro passo é escolher a polimerase apropriada. A polimerase Taq regular não pode ser revisada devido à falta de atividade da exonuclease 3'-5 'e a incompatibilidade reduzirá muito a eficiência de extensão dos fragmentos. Portanto, a polimerase Taq regular não pode amplificar com eficácia fragmentos alvo maiores que 5 kb. A polimerase Taq com modificação especial ou outra polimerase de alta fidelidade deve ser selecionada para melhorar a eficiência da extensão e atender às necessidades de amplificação de fragmentos longos. Além disso, a amplificação de fragmentos longos também requer o ajuste correspondente do desenho do primer, tempo de desnaturação, tempo de extensão, pH do tampão, etc. Normalmente, os primers com 18-24 bp podem levar a um melhor rendimento. A fim de evitar danos ao molde, o tempo de desnaturação a 94 ° C deve ser reduzido para 30 segundos ou menos por ciclo, e o tempo para aumentar a temperatura para 94 ° C antes da amplificação deve ser inferior a 1 minuto. Além disso, definir a temperatura de extensão em cerca de 68 ° C e projetar o tempo de extensão de acordo com a taxa de 1 kb / min pode garantir a amplificação eficaz de fragmentos longos.
A taxa de erro de amplificação por PCR pode ser reduzida usando várias DNA polimerases com alta fidelidade. Entre todas as Taq DNA polimerases encontradas até agora, a enzima Pfu tem a menor taxa de erro e a maior fidelidade (ver tabela anexa). Além da seleção da enzima, os pesquisadores podem reduzir ainda mais a taxa de mutação da PCR otimizando as condições de reação, incluindo a otimização da composição do tampão, a concentração da polimerase termoestável e a otimização do número do ciclo da PCR.
Categorias de produtos
PORQUE ESCOLHER-NOS
Desde a sua criação, nossa fábrica tem desenvolvido produtos de primeira classe com a adesão ao princípio
de qualidade primeiro. Nossos produtos ganharam excelente reputação na indústria e são valiosos e confiáveis entre clientes novos e antigos.








