FastKing gDNA Dissipando RT SuperMix
Recursos
■ Rápido: uma etapa para concluir a remoção do genoma e a transcrição reversa eficiente em 18 minutos, apenas adicionando modelos.
■ Alta eficiência: A transcriptase reversa é modificada com um motivo hidrofóbico, com eficiência de RT superior a 95%.
■ Simples e fácil: A DNase termossensível exclusiva tem efeito rápido, alta eficiência com tempo de reação mais curto e não afetará o cDNA.
Especificação
Modelo: Transcriptase reversa modificada por gene, gDNase
Procedimentos: Uma etapa (remoção de DNA genômico e RT)
Eficiência RT: > 95%
Modelo: 1 ng- 2 μg
Tempo de operação: ~ 18 min
Formulários: O cDNA transcrito reversamente pode ser usado em PCR convencional, PCR em tempo real, construção de biblioteca de cDNA, SAGE (Análise Serial de Expressão Gênica), extensão de iniciador e outros experimentos convencionais.
Todos os produtos podem ser personalizados para ODM / OEM. Para detalhes,clique em Serviço personalizado (ODM / OEM)
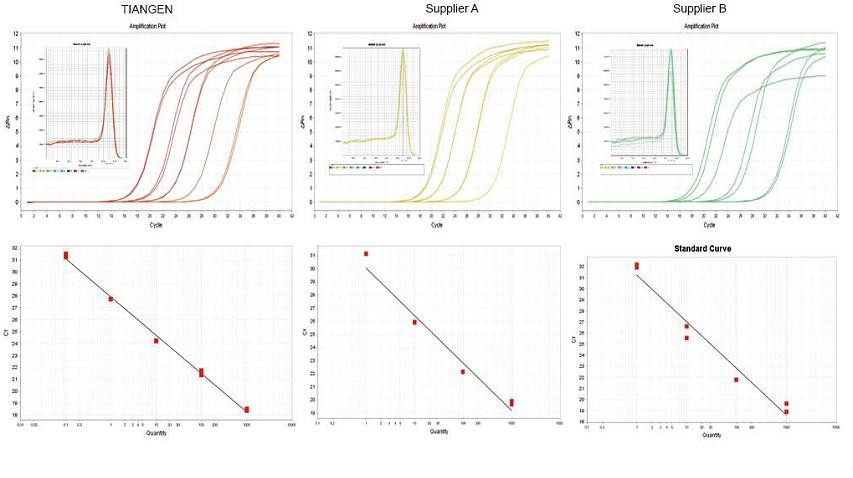 |
Exemplo experimental 1. O cDNA foi sintetizado usando reagentes quantitativos reversos de uma etapa de TIANGEN FastKing gDNA Dispelling RT SuperMix, produtos relevantes do Fornecedor A e Fornecedor B, respectivamente. Detecte o gene RN5 de camundongos usando TIANGEN Talent qPCR PreMix (SYBR Green), e a curva de amplificação, a curva de fusão e a curva padrão foram analisadas. Os resultados mostram que TIANGEN FastKing gDNA Dispelling RT SuperMix tem o valor Ct quantitativo mais alto após a transcrição reversa e excelente resistência ao estresse, e tem vantagens óbvias para modelos com resíduos de alta impureza. |
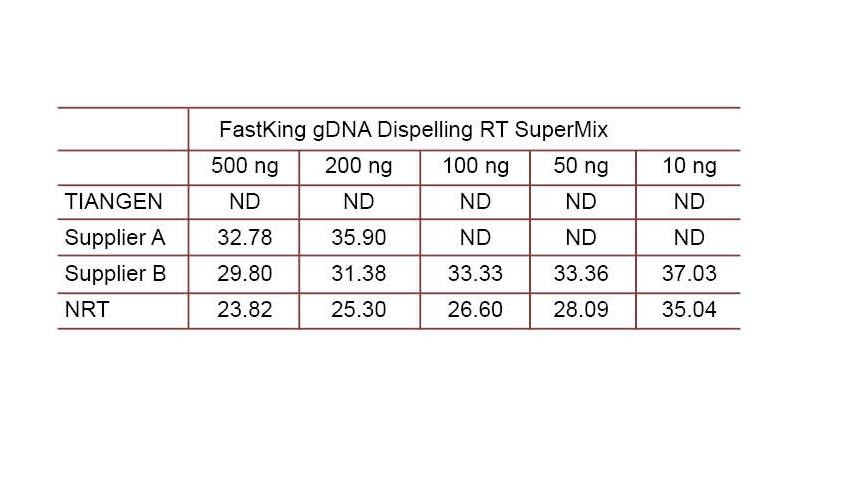 |
Exemplo experimental 2. O cDNA foi sintetizado usando reagentes quantitativos reversos de uma etapa da TIANGEN FastKing gDNA Dispelling RT SuperMix, produtos relevantes do Fornecedor A e Fornecedor B, respectivamente. Detecte o gene HsG humano usando TIANGEN Talent qPCR PreMix (SYBR Green) e adicione manualmente diferentes concentrações de DNA genômico para detectar a capacidade de remoção de gDNA de diferentes reagentes. Os resultados do Ct mostram que TIANGEN FastKing gDNA Dispelling RT SuperMix tem excelente capacidade para remover DNA genômico. Até 500 ng de resíduo de DNA genômico podem ser removidos perfeitamente sem afetar os resultados. ND: Não detectado. NRT: Detecção da mistura sem transcrição reversa. |
O RNA A-1 está degradado
—— Purificar RNA de alta qualidade sem contaminação. O material do qual o RNA é extraído deve ser o mais fresco possível para evitar a degradação do RNA. Analise a integridade do RNA em gel desnaturado antes da reação de RT. Após a extração do RNA, ele deve ser armazenado em formamida 100%. Se o inibidor de RNase for usado, a temperatura de aquecimento deve ser <45 ° C e o pH deve ser inferior a 8,0, caso contrário, o inibidor irá liberar toda a RNase ligada. Além disso, o inibidor de RNase deve ser adicionado em soluções contendo DTT ≥ 0,8 mM.
O RNA A-2 contém inibidores das reações de transcrição reversa
—— Os inibidores da transcrição reversa incluem SDS, EDTA, glicerol, pirofosfato de sódio, espermidina, formamida, sal de guanidina, etc. Misture o RNA de controle com a amostra e compare o rendimento com a reação de RNA de controle para verificar se há um inibidor. Lave a precipitação do RNA com etanol a 70% (v / v) para remover os inibidores.
A-3 emparelhamento insuficiente de primers usados para sintetizar a primeira fita de cDNA
—— Determine se a temperatura de recozimento é adequada para os primers usados no experimento. Para hexâmeros aleatórios, é recomendado manter a temperatura a 25 ° C por 10 min antes de atingir a temperatura de reação. Para primers específicos de gene (GSP), tente outro GSP ou mude para oligo (dT) ou hexâmero aleatório.
A-4 pequena quantidade de RNA inicial
——Aumente a quantidade de RNA. Para amostras de RNA menores que 50 ng, 0,1 μg a 0,5 μg de acetil BSA podem ser usados na síntese da primeira fita de cDNA
A-5 A sequência alvo não é expressa nos tecidos analisados.
——Experimente outros tecidos.
A reação de PCR A-6 falha
—— Para RT-PCR de duas etapas, o modelo de cDNA na etapa de PCR não pode exceder 1/5 do volume da reação.
A-1 recozimento não específico de primers e modelos
——A extremidade 3 'dos primers não deve conter 2-3 dG ou dC. Use primers específicos do gene na síntese da primeira fita em vez de primers aleatórios ou oligo (dT). Use uma temperatura de recozimento mais alta nos primeiros ciclos e, em seguida, uma temperatura de recozimento mais baixa. Use a Taq DNA polimerase hot-start para PCR para melhorar a especificidade da reação.
A-2 Projeto pobre de primers específicos de gene
—— Siga os mesmos princípios para o design do primer de amplificação.
RNA A-3 contaminado com DNA genômico
——Trate o RNA com DNase I de grau de PCR. Configure uma reação de controle sem transcrição reversa para detectar a contaminação do DNA.
A-4 Formação de dímero de primer
—— Projete primers sem sequências complementares na extremidade 3 '.
A-5 Mg muito alto2+ concentração
—— Otimize Mg2+ concentração para cada combinação de modelo e primer
A-6 contaminado com DNA estranho
—— Use pontas resistentes a aerossol e enzimas UDG.
A-1 O conteúdo do produto da primeira vertente é muito alto
—— Reduza a quantidade do produto da primeira fita na etapa de reação de PCR convencional.
Quantidade de primer A-2 muito alta na reação de PCR
——Reduzir a entrada do primer.
A-3 Muitos ciclos
——Otimizar as condições de reação PCR e reduzir o número do ciclo PCR.
A-4 Temperatura de recozimento muito baixa
—— Aumente a temperatura de recozimento para evitar iniciação e extensão não específicas.
A-5 Amplificação não específica de fragmentos de oligonucleotídeos gerados pela degradação de DNA por DNase - Extraia RNA de alta qualidade para evitar a contaminação de DNA.
RT-PCR é reverter transcrever RNA em cDNA, e então usar o cDNA reversamente transcrito como um modelo para a reação de PCR para amplificar o fragmento alvo. Escolha um dos primers aleatórios, Oligo dT e primers específicos do gene de acordo com as condições específicas do experimento. Todos os iniciadores acima podem ser usados para mRNA de células eucarióticas curtas sem estrutura em grampo.
Primer aleatório: Adequado para RNA longo com estrutura em grampo, bem como todos os tipos de RNA, como rRNA, mRNA, tRNA, etc. Eles são usados principalmente para reação RT-PCR de modelo único.
Oligo dT: Adequado para RNA com cauda PolyA (RNA procariótico, Oligo dT rRNA eucariótico e tRNA não têm caudas PolyA). Como o Oligo dT está ligado à cauda PolyA, a qualidade das amostras de RNA deve ser alta e mesmo uma pequena quantidade de degradação reduzirá muito a quantidade de síntese de cDNA de comprimento total.
Primer específico do gene: Complementar à sequência modelo, adequado para situações em que a sequência alvo é conhecida.
Existem duas maneiras:
1. Método de referência interna: Em teoria, cDNA são fragmentos de DNA de comprimentos diferentes, então o resultado da eletroforese é o esfregaço. Se a abundância de RNA for baixa, nenhum produto será mostrado na eletroforese, mas isso não significa que nenhum produto será amplificado por PCR. Em geral, a referência interna pode ser usada para detectar cDNA. Se a referência interna apresentar resultados, a qualidade do cDNA pode ser basicamente garantida (em alguns casos, se o fragmento do gene alvo for muito longo, pode haver exceções).
2. Se houver um gene conhecido amplificado por esse modelo, ele pode ser verificado pelos primers desse gene. A amplificação da referência interna não significa necessariamente que não haja nenhum problema com o cDNA. Como a referência interna tem alta abundância em cDNA, é fácil de amplificar. Se o cDNA for parcialmente degradado por várias razões, do ponto de vista da probabilidade, os resultados de PCR de genes alvo de baixa abundância serão grandemente afetados. Embora a referência interna ainda seja alta em abundância, a amplificação provavelmente não será afetada.
Degrada parcialmente o RNA. Detectar a integridade e purificar o RNA
Os conteúdos de RNA de diferentes espécies podem ser diferentes, mas em geral, o RNA total extraído deve conter duas bandas 28S e 18S claras em eletroforese em gel, e o brilho da primeira banda deve ser duas vezes maior que o da última. A banda 5S indica que o RNA foi degradado e seu brilho é proporcional ao grau de degradação. O sucesso da amplificação da referência interna não significa que não haja nenhum problema com o RNA, porque a referência interna está em alta abundância, o RNA pode ser amplificado desde que a degradação não seja severa. O OD260/ OD280a proporção de RNA puro medido por espectrofotômetro deve estar entre 1,9 e 2,1. Uma pequena quantidade de impureza de proteína no RNA reduzirá a proporção. Contanto que o valor não seja muito baixo, o RT não será afetado. O que mais importa para a RT é a integridade do RNA.
A extensão do gene de referência interna pode apenas indicar que a RT foi bem-sucedida, mas não está necessariamente relacionada à qualidade da fita de cDNA. Como os fragmentos de referência internos são geralmente pequenos em tamanho e alta expressão, eles são mais fáceis de serem bem-sucedidos na transcrição reversa. No entanto, o tamanho e a expressão do gene alvo variam de gene para gene. A qualidade do cDNA não pode ser avaliada apenas por referência interna, especialmente para os fragmentos alvo com mais de 2 kb.
Algumas amostras têm estruturas secundárias complexas, ou têm um rico conteúdo de GC, ou são preciosas com baixa abundância. Nestes casos, a transcriptase reversa apropriada deve ser selecionada de acordo com o tamanho do fragmento alvo e da amostra. Para modelos de RNA com alto conteúdo de GC e estrutura secundária complexa, é difícil abrir a estrutura secundária em baixa temperatura ou com transcriptase reversa comum. Para esses modelos, Quant Reverse Transcriptase pode ser selecionada, uma vez que seu desempenho de transcrição reversa é obviamente melhor do que o da transcriptase reversa da série M-MLV, que pode reverter a transcrição de vários modelos de RNA de forma eficiente e transcrever o RNA na primeira fita de cDNA na extensão máxima. Ao usar o kit geral de transcriptase reversa, o sistema de 20 μl só pode reverter efetivamente a transcrição de 1 μg de RNA total. Preste atenção à capacidade máxima de RT do kit. Se o modelo for adicionado em excesso, a transcrição reversa favorecerá o RNA com alta abundância. Portanto, é melhor não exceder a capacidade máxima do sistema.
A-1 Determine se o RNA está gravemente degradado e se a RT é bem-sucedida
Em geral, a razão para a falha da amplificação de referência interna é freqüentemente causada por uma grave degradação do RNA. Outra razão possível é a falha da transcrição reversa. A referência interna não pode ser usada como um padrão para julgar a qualidade da fita simples de cDNA, mas pode ser usada como um padrão para julgar se a transcrição reversa é bem-sucedida se não houver nenhum problema com a qualidade do RNA. O mais importante no processo de transcrição reversa é manter uma temperatura constante e um sistema de reação constante para melhorar a eficiência da reação.
A-2 Determine se os primers para amplificar genes de referência internos são confiáveis e se há algum problema com os reagentes usados na PCR.
Para quantificação relativa, o RNA deve ser quantificado antes da transcrição reversa, que também é necessária em muitos kits de transcrição reversa, por exemplo, quantificar a entrada de RNA como 1 μg. Uma vez que o cDNA transcrito reversamente é uma solução mista, incluindo RNA, oligo dT, enzima, dNTP e até mesmo um pequeno resíduo de DNA, o desvio será causado, então é impossível quantificar com precisão o cDNA. Portanto, a quantificação de RNA é necessária. Considerando que a eficiência da transcrição reversa é a mesma entre as diferentes amostras, a quantidade de cDNA obtida deve ser a mesma, e a análise quantitativa pode mostrar a comparação dos níveis de expressão de diferentes genes na mesma quantidade de RNA total. Ao realizar PCR quantitativo de fluorescência relativa, o cDNA quantitativo pode não ser necessário após a transcrição reversa porque o gene de referência interno pode atuar como referência.
Está relacionado principalmente aos genes, e a transcrição reversa de fragmentos longos não é viável para a maioria dos genes. Em primeiro lugar, a eficiência da transcrição reversa é muito menor do que a da PCR. Em segundo lugar, a região rica em GC e a estrutura secundária de muitos genes restringem a transcrição reversa e a PCR. Finalmente, a fidelidade e a eficiência da amplificação da PCR são difíceis de garantir ao mesmo tempo. No processo de transcrição reversa, ninguém pode garantir a obtenção de fragmentos longos para genes de baixa cópia, especialmente usando oligo dT. Já para 5 'UTR com mais GC, é ainda mais difícil. Portanto, ainda é um método razoável para reverter a transcrição com primers aleatórios, encontrar os locais de clivagem natural no fragmento alvo, amplificar por segmentos e, em seguida, realizar a digestão de restrição e a ligação. Em geral, é difícil amplificar diretamente fragmentos maiores que 2 kb, mas nem sempre é impossível obter: 1. Em primeiro lugar, garantir a integridade do RNA / mRNA, preferindo-se a extração de TRIZOL. 2. O kit RT-PCR M-MLV pode ser usado diretamente. Prolongue o tempo de recozimento e aumente o número do ciclo no processo de amplificação de maneira adequada. Alternativamente, a PCR interna pode ser aplicada, ou realizar uma ou duas reações primeiro com desnaturação apropriadamente prolongada e tempo de extensão antes da amplificação por PCR normal, o que pode ajudar a estender fragmentos. Preste atenção à fidelidade da polimerase. 3.Long Taq pode ser usado em PCR para obter resultados ideais. 4.Para aplicação de expressão de proteína, polimerase de alta fidelidade deve ser aplicada.
Existem dois tipos de transcriptase reversa oferecidos pela TIANGEN: Quant / King RTase e TIANScript M-MLV. A principal diferença entre eles é a quantidade de entrada de modelos. Quant é uma transcriptase reversa única, que é diferente do comumente usado M-MLV derivado do vírus da leucemia murina de Moloney. Quant é uma nova transcriptase reversa de alta eficiência expressa de forma recombinante pela engenharia de Escherichia coli. Quant é adequado para amplificar 50 ng-2 μg de RNA com alta atividade de transcrição reversa e alto rendimento. Comparado com MMLV ou AMV comum, a maior característica de Quant é que ele tem uma afinidade muito forte com modelos de RNA e pode reverter modelos complexos de transcrição sem desnaturação de alta temperatura. Para modelos com maior conteúdo de GC, a eficiência reversa é maior. No entanto, esta transcriptase reversa tem atividade RNase H, que pode afetar o comprimento do produto de cDNA (adequado para modelos <4,5 kb). Para a transcrição reversa convencional, a transcriptase reversa TIANScript MMLV é recomendada. Esta RTase é uma enzima modificada com atividade RNase H muito fraca, que é adequada para síntese de cDNA longa (> 5 kb).
A transcrição reversa de uma etapa e a amplificação por PCR são concluídas no mesmo tubo, sem abrir a tampa do tubo entre a síntese de cDNA e a amplificação, o que é útil para reduzir a contaminação. Como todas as amostras de cDNA obtidas são utilizadas para amplificação, a sensibilidade é maior, com um mínimo de 0,01 pg de RNA total. Para RTPCR de uma etapa bem-sucedido, iniciadores específicos de gene são geralmente usados para iniciar a síntese de cDNA. O método de duas etapas, ou seja, a transcrição reversa e a amplificação por PCR é realizada em duas etapas. Em primeiro lugar, a transcrição reversa é realizada a partir de um molde de RNA para obter cDNA, e o cDNA obtido é submetido a uma ou mais reações de PCR diferentes. O método de duas etapas pode usar oligo (dT) ou iniciadores aleatórios para guiar a síntese da primeira fita de cDNA e pode fazer a transcrição reversa de todas as informações de mRNA de uma amostra específica.
Categorias de produtos
PORQUE ESCOLHER-NOS
Desde a sua criação, nossa fábrica tem desenvolvido produtos de primeira classe com a adesão ao princípio
de qualidade primeiro. Nossos produtos ganharam excelente reputação na indústria e são valiosos e confiáveis entre clientes novos e antigos.