2 × Pfu PCR Mix
Definição de Atividade
A atividade de 1 unidade (U) Pfu DNA Polimerase é definida como a quantidade de enzima necessária para incorporar 10 nmol de desoxinucleotídeos em substâncias insolúveis em ácido a 74 ° C em 30 min usando DNA de esperma de salmão ativado como molde / iniciador.
Controle de qualidade
A pureza pela detecção de SDS-PAGE é superior a 99%; Nenhuma atividade de nuclease exógena é detectada; O gene de cópia única no genoma humano pode ser amplificado de forma eficaz; Nenhuma mudança significativa na atividade quando armazenado em temperatura ambiente por uma semana.
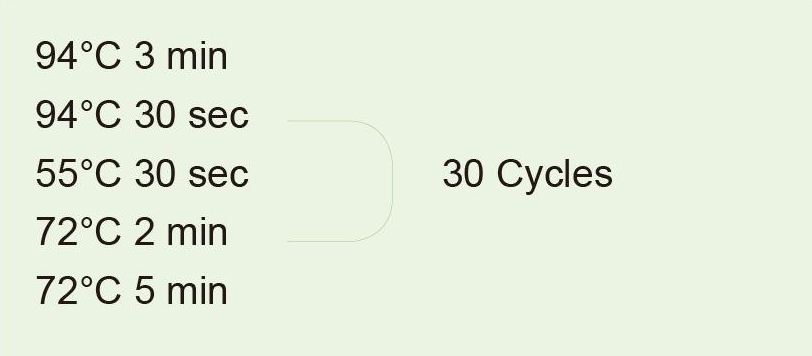
Parâmetros Técnicos Principais
Tem atividade de exonuclease 3′-5 ′ e nenhuma atividade de exonuclease 5′-3 ′. A velocidade de extensão da amplificação do DNA é inferior à da Taq polimerase e, geralmente, a velocidade de extensão da enzima Pfu é de 0,5-1 kb por minuto. A estabilidade térmica do Pfu é melhor do que o Taq. Para modelos com alto teor de GC, a temperatura de desnaturação pode ser aumentada para 98 ° C, o que não tem efeito sobre a atividade da polimerase Pfu. O produto de PCR é de extremidade cega, que pode ser adicionado com saliências de 3'-dA antes de ser ligado ao vetor TA ou clonado com vetor de extremidade cega
Formulários
Ele pode ser usado para amplificação de alta fidelidade de DNA, como clonagem de expressão gênica, mutação dirigida ao local, análise de polimorfismo de nucleotídeo único (SNP) e reparo de extremidade.
Todos os produtos podem ser personalizados para ODM / OEM. Para detalhes,clique em Serviço personalizado (ODM / OEM)
  |
Use DNA genômico como molde para amplificar o fragmento de 1kb. Após a reação de PCR, leve 5 μl para detecção de eletroforese. |
Modelo A-1
■ O modelo contém impurezas de proteínas ou inibidores Taq, etc. —— Purifique o modelo de DNA, remova as impurezas de proteínas ou extraia o DNA do modelo com kits de purificação.
■ A desnaturação do modelo não está completa —— Aumente apropriadamente a temperatura de desnaturação e prolongue o tempo de desnaturação.
■ Degradação do modelo ——Reprepare o modelo.
A-2 Primer
■ Baixa qualidade dos primers —— Sintetize novamente o primer.
■ Degradação do primer —— Aliquote os primers de alta concentração em um pequeno volume para preservação. Evite congelamento e descongelamento múltiplo ou criopreservação de 4 ° C por longo prazo.
■ Projeto inadequado de primers (por exemplo, o comprimento do primer não é suficiente, dímero formado entre os primers, etc.) -Redesign primers (evita a formação de primer dímero e estrutura secundária)
A-3 Mg2+concentração
■ Mg2+ a concentração está muito baixa —— Aumente o Mg de maneira adequada2+ concentração: Otimize o Mg2+ concentração por uma série de reações de 1 mM a 3 mM com um intervalo de 0,5 mM para determinar o Mg ideal2+ concentração para cada molde e primer.
Temperatura de recozimento A-4
■ A alta temperatura de recozimento afeta a ligação do primer e do template. ——Reduza a temperatura de recozimento e otimize a condição com um gradiente de 2 ° C.
Tempo de extensão A-5
■ Tempo de extensão curto —— Aumente o tempo de extensão.
Fenômenos: Amostras negativas também mostram as bandas da sequência alvo.
Contaminação A-1 de PCR
■ Contaminação cruzada da sequência-alvo ou produtos de amplificação ——Cuidado para não pipetar a amostra que contém a sequência-alvo na amostra negativa ou derramar para fora do tubo de centrífuga. Os reagentes ou equipamentos devem ser autoclavados para eliminar os ácidos nucléicos existentes, e a existência de contaminação deve ser determinada por meio de experimentos de controle negativo.
■ Contaminação do reagente —— Aliquote os reagentes e armazene-os em temperatura baixa.
A-2 Primer
■ Mg2+ a concentração está muito baixa —— Aumente o Mg de maneira adequada2+ concentração: Otimize o Mg2+ concentração por uma série de reações de 1 mM a 3 mM com um intervalo de 0,5 mM para determinar o Mg ideal2+ concentração para cada molde e primer.
■ Projeto de primer impróprio e a sequência alvo tem homologia com a sequência não alvo. —— Refaça os primers.
Fenômenos: As bandas de amplificação de PCR são inconsistentes com o tamanho esperado, seja grande ou pequeno, ou às vezes ocorrem bandas de amplificação específicas e bandas de amplificação não específicas.
A-1 Primer
■ Baixa especificidade do primer
—— Primer de redesenho.
■ A concentração do primer está muito alta —— Aumente adequadamente a temperatura de desnaturação e prolongue o tempo de desnaturação.
A-2 Mg2+ concentração
■ O Mg2+ a concentração é muito alta —— Reduza adequadamente a concentração de Mg2 +: Otimize o Mg2+ concentração por uma série de reações de 1 mM a 3 mM com um intervalo de 0,5 mM para determinar o Mg ideal2+ concentração para cada molde e primer.
Polimerase A-3 Termostável
■ Quantidade excessiva de enzima - reduza a quantidade de enzima de forma adequada em intervalos de 0,5 U.
Temperatura de recozimento A-4
■ A temperatura de recozimento está muito baixa —— Aumente apropriadamente a temperatura de recozimento ou adote o método de recozimento de dois estágios
Ciclos de PCR A-5
■ Muitos ciclos de PCR —— Reduza o número de ciclos de PCR.
A-1 Primer—— Especificidade pobre —— Desenhe novamente o primer, mude a posição e o comprimento do primer para aumentar sua especificidade; ou realizar PCR aninhado.
DNA de modelo A-2
——O modelo não é puro —— Purifique o modelo ou extraia o DNA com kits de purificação.
A-3 Mg2+ concentração
——Mg2+ a concentração é muito alta —— Reduza o Mg de maneira adequada2+ concentração: Otimize o Mg2+ concentração por uma série de reações de 1 mM a 3 mM com um intervalo de 0,5 mM para determinar o Mg ideal2+ concentração para cada molde e primer.
A-4 dNTP
—— A concentração de dNTPs está muito alta —— Reduza a concentração de dNTP de forma adequada
Temperatura de recozimento A-5
—— Temperatura de recozimento muito baixa —— Aumente apropriadamente a temperatura de recozimento
Ciclos A-6
—— Muitos ciclos —— Otimize o número do ciclo

O primeiro passo é escolher a polimerase apropriada. A polimerase Taq regular não pode ser revisada devido à falta de atividade da exonuclease 3'-5 'e a incompatibilidade reduzirá muito a eficiência de extensão dos fragmentos. Portanto, a polimerase Taq regular não pode amplificar com eficácia fragmentos alvo maiores que 5 kb. A polimerase Taq com modificação especial ou outra polimerase de alta fidelidade deve ser selecionada para melhorar a eficiência da extensão e atender às necessidades de amplificação de fragmentos longos. Além disso, a amplificação de fragmentos longos também requer o ajuste correspondente do desenho do primer, tempo de desnaturação, tempo de extensão, pH do tampão, etc. Normalmente, os primers com 18-24 bp podem levar a um melhor rendimento. A fim de evitar danos ao molde, o tempo de desnaturação a 94 ° C deve ser reduzido para 30 segundos ou menos por ciclo, e o tempo para aumentar a temperatura para 94 ° C antes da amplificação deve ser inferior a 1 minuto. Além disso, definir a temperatura de extensão em cerca de 68 ° C e projetar o tempo de extensão de acordo com a taxa de 1 kb / min pode garantir a amplificação eficaz de fragmentos longos.
A taxa de erro de amplificação por PCR pode ser reduzida usando várias DNA polimerases com alta fidelidade. Entre todas as Taq DNA polimerases encontradas até agora, a enzima Pfu tem a menor taxa de erro e a maior fidelidade (ver tabela anexa). Além da seleção da enzima, os pesquisadores podem reduzir ainda mais a taxa de mutação da PCR otimizando as condições de reação, incluindo a otimização da composição do tampão, a concentração da polimerase termoestável e a otimização do número do ciclo da PCR.
Categorias de produtos
PORQUE ESCOLHER-NOS
Desde a sua criação, nossa fábrica tem desenvolvido produtos de primeira classe com a adesão ao princípio
de qualidade primeiro. Nossos produtos ganharam excelente reputação na indústria e são valiosos e confiáveis entre clientes novos e antigos.








